PubMed API
PubMed検索APIについての公式ドキュメントは
E-utilities Quick Start - Entrez Programming Utilities Help - NCBI Bookshelf
にあります。
http://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi?db=pubmed&term=<query>
の<query>部分に検索したい単語を入れてアクセスするのが基本的な使い方です。以下にcurlで叩く例を示します。
デフォルトのフォーマットはXMLですが今回はJSONで受け取りたいので、retmodeパラメータにjsonを指定します。
結果がJSONで帰ってきたのがわかります。ちなみにprogressを表示しないようcurlに-sオプションを付けています。
jq
さて、この結果JSONをRubyで取り扱う... 前に、jqという便利なツールを紹介します。
macであれば Homebrew を使って以下のようにインストールできます。
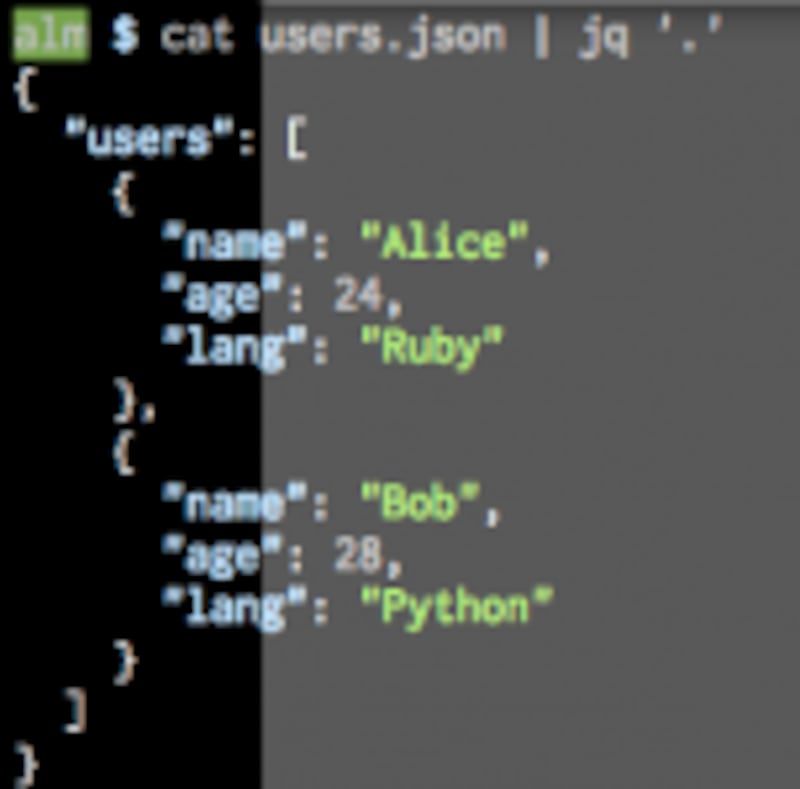
jqのよくある使い方は、JSONを標準入力で受け取ってフィルタリングするというものです。たとえば以下の様なusers.jsonというファイルがあったとして、
これをcat users.json | jq '.'とすれば整形して出力されます。
jqは様々なフィルタリング機能を備えており、特定のフィールドのみ出力することや
条件式でデータを選択することができます。
sedやawk, grepとは異なる独自の文法を持っているため学習コストがかかるのが面倒といえば面倒ですが、コマンドラインでJSONデータをちょっと検索したり切り出したりする時に便利です。
このjqを使うと、先ほどのPubMed検索結果JSONから文献idのみを抽出するのも簡単です。
それでは、次のページでRubyを使ってAPIを叩き、JSONをHashオブジェクトに変換する方法を見ていきます。